Pythonで化学構造式を扱うためには、主にRDKitというライブラリが使用されます。
RDKitは化学情報学のための強力なツールで、分子の生成、操作、解析、視覚化などさまざまな機能を提供しています。
ここではRDKitを用いて化学構造式を扱う基本的なステップについて解説します。
利用準備
まずは利用開始までに行うことと知っておくべき知識を解説します。
インストール
まずはRDKitをインストールします。
pip install rdkit-pypi3Dモデルも作りたい場合はpy3Dmolもインストールしておきます。
pip install py3Dmol分子の記述方法(SMILES)
RDKitを使うと、SMILES(Simplified Molecular Input Line Entry System)表記や他の形式を用いて分子を生成できます。
SMILESは分子構造を簡潔な文字列形式で表現する方法です。
いくつか例を示します。
- メタン:C
- プロパン:CCC
- エタノール:CCO
- アセチレン:C#C
- クロロプレンモノマー:C=CC(Cl)=C
- ベンゼン:C1=CC=CC=C1
- アセチルサリチル酸:CC(=O)OC1=CC=CC=C1C(=O)O
現在ではデータ解析の分野で分子を数値化するために活用されています。
分子の構造式を画像で取得する
まずは分子を生成して画像として取得します。
分子の生成
まずはエタノール分子を生成してみます。
RDKitのChemモジュールからMolFromSmiles関数を使用して、SMILES表記から分子オブジェクトを生成します。
from rdkit import Chem
# SMILES表記で分子を定義
smiles = 'CCO' # エタノールの例
# 分子オブジェクトの生成
molecule = Chem.MolFromSmiles(smiles)以下のように分かりやすいエタノール分子を作ってくれました。
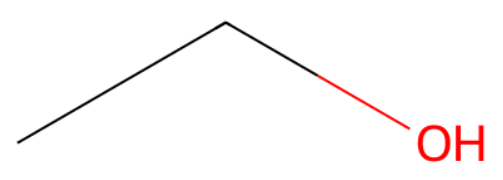
分子の構造式の描画
生成した分子オブジェクトは、Drawモジュールを使用して視覚化することができます。
from rdkit.Chem import Draw
# 分子の構造式を描画
image = Draw.MolToImage(molecule)
# 画像を表示
image.show()image.show() メソッドを使用した場合、画像は自動的にはどこにも保存されません。
このメソッドは画像を直接画面に表示するためのものです。
画像を保存するには、save() メソッドを使用してファイルに書き込む必要があります。
image_path = 'path/to/your/ethanol.png' # 保存先のファイルパスを設定
image.save(image_path) # 画像をファイルに保存Pythonプログラムと同じフォルダに保存する場合は保存したい画像のファイル名だけでも大丈夫です。
image.save("ethanol.png") # 画像をファイルに保存3D構造の分子を描画する
AllChem.EmbedMoleculeメソッドを使って分子の3D構造を生成します。
まず描画するためにpy3Dmolをインストールします。
pip install py3Dmolライブラリをインポートします。
from rdkit import Chem
from rdkit.Chem import AllChem
import py3Dmolエタノール分子をSMILESで記述します。
# 分子の生成と3D構造の準備
mol = Chem.MolFromSmiles('CCO')次に分子に水素原子を追加しています。
多くのSMILES表記では水素が省略されているため、これにより完全な3D構造を生成するための準備をしています。
mol = Chem.AddHs(mol)次に分子の3D座標を生成します。
AllChem.EmbedMoleculeはランダムな3D構造を生成します。
AllChem.ETKDGは3D構造生成のためのアルゴリズムを指定しています。
更にAllChem.UFFOptimizeMoleculeで分子の3D構造をエネルギー最小化アルゴリズムを用いて最適化しています。
これにより、より現実的な3D構造が得られます。
AllChem.EmbedMolecule(mol, AllChem.ETKDG())
AllChem.UFFOptimizeMolecule(mol)次に分子の3D構造情報をMolBlock形式(テキストベースの形式)に変換します。
これはpy3Dmolで使用するための準備です。
# 分子の3D構造データの取得
mb = Chem.MolToMolBlock(mol)最後に3Dモデルを表示します。
# py3Dmolを使用して3D表示
view = py3Dmol.view(width=400, height=400) # ビューアーサイズ
view.addModel(mb, 'mol') # ビューアーにデータ追加、molはデータ形式
view.setStyle({'stick': {}}) # 分子の表示スタイルを設定
view.zoomTo() # 分子がビューア内で適切に表示されるようにズーム調整
view.show()エタノールの3Dモデルが生成されました。
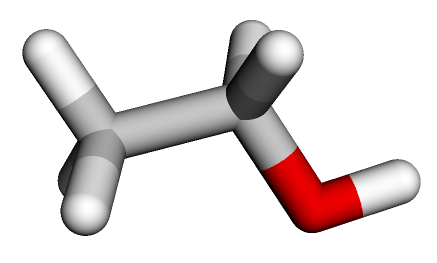
Jupyter Notebookの実行環境では、その場で回転させることも出来ます。
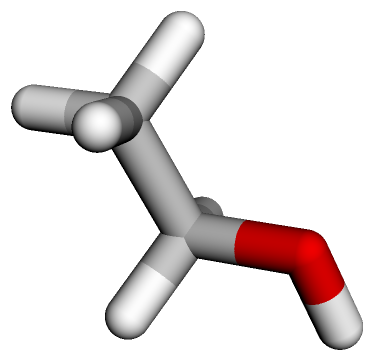
分子の情報を得る
生成した分子から原子の数や結合の種類、分子量など様々な情報を取得してみます。
同様にエタノールを題材にします。
from rdkit import Chem
# SMILES表記で分子を定義
smiles = 'CCO' # エタノールの例
# 分子オブジェクトの生成
molecule = Chem.MolFromSmiles(smiles)原子と結合の情報
GetNumAtoms()メソッドは、その分子内にある原子の総数を返します。
水素原子はカウントされません。
# 原子の数を取得
num_atoms = molecule.GetNumAtoms()エタノールの場合3が返されます。
どのような原子が含まれているか確認するには以下のコードを実行します。
# 各原子についての情報を取得
for atom in molecule.GetAtoms():
print(atom.GetSymbol()) # 原子記号"CCO"と出力されます。
エタノール分子の結合数を確認するにはGetNumBonds()メソッドを使います。
注意点として、GetNumBonds() メソッドは単純な結合の数を返します。つまり、二重結合や三重結合も単一結合としてカウントされます。
また水素原子と他の原子との結合はカウントされません。
# 結合に関する情報
num_bonds = molecule.GetNumBonds()エタノールの場合2が出力され、CH3-CH2結合、CH2-OH結合が計数の対象です。
分子量の計算
rdMolDescriptorsモジュールを使用して分子量を計算できます。
from rdkit.Chem import rdMolDescriptors
# 分子量の計算
molecular_weight = rdMolDescriptors.CalcExactMolWt(molecule)エタノールの場合、"46.041864812"と出力され、一致しました。
参考資料
オススメ書籍
・化学のためのPythonによるデータ解析・機械学習入門
データ分析に必要な最低限の知識を解説したうえで、化学プラントで得られるデータの扱い方が紹介されています。
脱ブタン塔や排煙脱硝装置を例に取り上げられておりイメージしやすくなっています。
-

-
化学のためのPythonによるデータ解析・機械学習入門
www.amazon.co.jp
・Pythonによる時系列分析: 予測モデル構築と企業事例
プロセス製造において時系列データの分析は欠かせません。
どのように時系列予測モデルを構築し、ビジネスへ活用していくかを詳細なPythonコードとともに解説してくれます。
-

-
Pythonによる時系列分析: 予測モデル構築と企業事例
www.amazon.co.jp
・PyCaretで学ぶ 機械学習入門
機械学習モデルを構築するのは想像以上に手間がかかります。
その一連の作業を自動化できるPyCaretというライブラリの使い方が分かりやすく解説されています。
-

-
PyCaretで学ぶ 機械学習入門
www.amazon.co.jp

